Kit TIANcombi DNA Lyse & Det PCR
Características
■ Sencillo y rápido: el ADN de diferentes tejidos se puede extraer en 5 minutos sin necesidad de triturar nitrógeno líquido.
■ Amplias aplicaciones: aplicable a hojas de plantas, semillas, tejidos animales, muestras de sangre (sangre fresca, anticoagulación, coágulos de sangre, manchas de sangre seca, etc.), levaduras y bacterias.
■ Gran compatibilidad: el reactivo de PCR es adecuado para la amplificación de ADN extraído de diversas fuentes de muestras.
Aplicaciones
■ Detección de genes: opción ideal para la detección de genes a gran escala.
Notas importantes
■ Para muestras que contienen un alto nivel de fenoles, como hojas de algodón, la cantidad de entrada de la muestra debe ser estrictamente inferior a 0,4 mg; de lo contrario, la reacción de PCR se verá afectada.
Todos los productos se pueden personalizar para ODM / OEM. Para detalles,haga clic en Servicio personalizado (ODM / OEM)
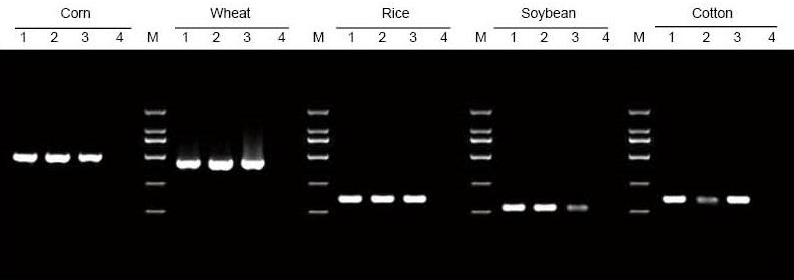 |
Se extrajo ADN de 5 mg de hojas y semillas de maíz, trigo, arroz, soja y algodón, respectivamente. El ADN se amplificó mediante PCR utilizando cebadores específicos. Se cargaron 6 µl de ADN del total de 20 µl de eluyentes por carril. 1: genoma de control positivo; 2: dejar muestras; 3: muestras de semillas; 4: NTC; 5: imprimaciones D2000 |
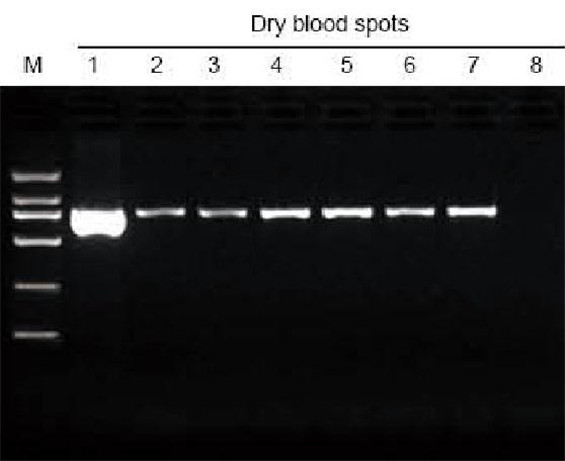 |
M: marcador TIANGEN D2000; 1: control positivo; 2-7: El número de manchas de sangre seca en el papel de filtro es 1-6 respectivamente; 8: Control negativo. El punzón de 3 mm se utilizó para tomar las manchas de sangre seca del papel de filtro como material para la prueba de extracción. Se cargaron 6 µl de ADN del total de 20 µl de eluyentes por carril. |
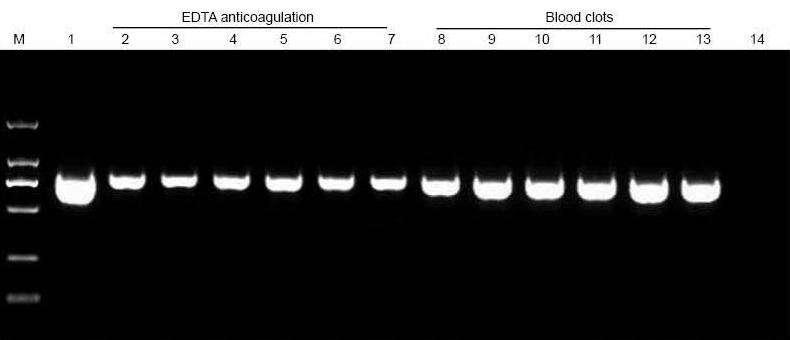 |
M: marcador TIANGEN D2000; 1: Control positivo (se utilizó ADN genómico como molde); 2-7: Las cantidades de sangre añadidas son 10 µl, 20 µl, 30 µl, 40 µl, 50 µl y 60 µl, respectivamente; 8-13: Las cantidades de sangre añadidas son 10 µl, 20 µl, 30 µl, 40 µl, 50 µl y 60 µl, respectivamente; 14: NTC. Se cargaron 6 µl de ADN del total de 20 µl de eluyentes en el gel de agarosa. |
Plantilla A-1
■ La plantilla contiene impurezas de proteínas o inhibidores de Taq, etc. —Purifique la plantilla de ADN, elimine las impurezas de proteínas o extraiga el ADN de la plantilla con kits de purificación.
■ La desnaturalización de la plantilla no es completa —— Aumente adecuadamente la temperatura de desnaturalización y prolongue el tiempo de desnaturalización.
■ Degradación de la plantilla: vuelva a preparar la plantilla.
Imprimación A-2
■ Mala calidad de los cebadores: vuelva a sintetizar el cebador.
■ Degradación de la imprimación: alícuota las imprimaciones de alta concentración en un volumen pequeño para su conservación. Evite la congelación y descongelación múltiples o criopreservados a 4 ° C a largo plazo.
■ Diseño inadecuado de los cebadores (por ejemplo, la longitud del cebador no es suficiente, se forma un dímero entre los cebadores, etc.) - Rediseñe los cebadores (evite la formación del dímero del cebador y la estructura secundaria)
A-3 Mg2+concentración
■ Mg2+ la concentración es demasiado baja —— Aumente adecuadamente el Mg2+ concentración: Optimice el Mg2+ concentración mediante una serie de reacciones de 1 mM a 3 mM con un intervalo de 0,5 mM para determinar el Mg óptimo2+ concentración para cada plantilla y cebador.
A-4 Temperatura de recocido
■ La alta temperatura de recocido afecta la unión del cebador y la plantilla. ——Reducir la temperatura de recocido y optimizar la condición con un gradiente de 2 ° C.
A-5 Tiempo de extensión
■ Tiempo de extensión corto —— Aumente el tiempo de extensión.
Fenómenos: las muestras negativas también muestran las bandas de la secuencia objetivo.
A-1 Contaminación de PCR
■ Contaminación cruzada de la secuencia diana o los productos de amplificación —— Tenga cuidado de no pipetear la muestra que contiene la secuencia diana en la muestra negativa ni derramarla fuera del tubo de centrífuga. Los reactivos o el equipo deben esterilizarse en autoclave para eliminar los ácidos nucleicos existentes, y la existencia de contaminación debe determinarse mediante experimentos de control negativo.
■ Contaminación de los reactivos ——Aliquiar los reactivos y almacenar a baja temperatura.
A-2 Primer
■ Mg2+ la concentración es demasiado baja —— Aumente adecuadamente el Mg2+ concentración: Optimice el Mg2+ concentración mediante una serie de reacciones de 1 mM a 3 mM con un intervalo de 0,5 mM para determinar el Mg óptimo2+ concentración para cada plantilla y cebador.
■ Diseño de cebador inadecuado y la secuencia objetivo tiene homología con la secuencia no objetivo. —— Rediseño de imprimaciones.
Fenómenos: Las bandas de amplificación por PCR no son coherentes con el tamaño esperado, ya sea grande o pequeño, o en ocasiones se producen tanto bandas de amplificación específicas como bandas de amplificación no específicas.
Imprimación A-1
■ Pobre especificidad del cebador
—— Imprimación de rediseño.
■ La concentración de imprimación es demasiado alta —— Aumente adecuadamente la temperatura de desnaturalización y prolongue el tiempo de desnaturalización.
A-2 Mg2+ concentración
■ El Mg2+ la concentración es demasiado alta —— Reduzca adecuadamente la concentración de Mg2 +: Optimice la concentración de Mg2+ concentración mediante una serie de reacciones de 1 mM a 3 mM con un intervalo de 0,5 mM para determinar el Mg óptimo2+ concentración para cada plantilla y cebador.
Polimerasa termoestable A-3
■ Cantidad excesiva de enzimas: reduzca la cantidad de enzimas de manera adecuada a intervalos de 0,5 U.
A-4 Temperatura de recocido
■ La temperatura de recocido es demasiado baja —— Aumente adecuadamente la temperatura de recocido o adopte el método de recocido de dos etapas
Ciclos de PCR A-5
■ Demasiados ciclos de PCR: reduzca el número de ciclos de PCR.
Imprimación A-1—— Escasa especificidad —— Rediseñe el cebador, cambie la posición y la longitud del cebador para mejorar su especificidad; o realizar una PCR anidada.
Plantilla de ADN A-2
——La plantilla no es pura ——Purifique la plantilla o extraiga el ADN con kits de purificación.
A-3 Mg2+ concentración
——Mg2+ la concentración es demasiado alta —— Reduzca adecuadamente el Mg2+ concentración: Optimice el Mg2+ concentración mediante una serie de reacciones de 1 mM a 3 mM con un intervalo de 0,5 mM para determinar el Mg óptimo2+ concentración para cada plantilla y cebador.
A-4 dNTP
——La concentración de dNTP es demasiado alta —— Reducir la concentración de dNTP de forma adecuada
A-5 Temperatura de recocido
——Temperatura de recocido demasiado baja —— Aumente adecuadamente la temperatura de recocido
Ciclos A-6
——Demasiados ciclos ——Optimizar el número de ciclo

El primer paso es elegir la polimerasa adecuada. La polimerasa Taq regular no se puede corregir debido a la falta de actividad exonucleasa 3'-5 ', y el desajuste reducirá en gran medida la eficacia de extensión de los fragmentos. Por lo tanto, la polimerasa Taq regular no puede amplificar eficazmente fragmentos diana mayores de 5 kb. Se debe seleccionar la polimerasa Taq con modificación especial u otra polimerasa de alta fidelidad para mejorar la eficiencia de extensión y satisfacer las necesidades de amplificación de fragmentos largos. Además, la amplificación de fragmentos largos también requiere el ajuste correspondiente del diseño del cebador, el tiempo de desnaturalización, el tiempo de extensión, el pH del tampón, etc. Normalmente, los cebadores con 18-24 pb pueden conducir a un mejor rendimiento. Para evitar daños en la plantilla, el tiempo de desnaturalización a 94 ° C debe reducirse a 30 segundos o menos por ciclo, y el tiempo para aumentar la temperatura a 94 ° C antes de la amplificación debe ser inferior a 1 min. Además, establecer la temperatura de extensión en aproximadamente 68 ° C y diseñar el tiempo de extensión de acuerdo con la velocidad de 1 kb / min puede garantizar una amplificación eficaz de fragmentos largos.
La tasa de error de la amplificación por PCR se puede reducir utilizando varias ADN polimerasas con alta fidelidad. Entre todas las ADN polimerasas Taq encontradas hasta ahora, la enzima Pfu tiene la tasa de error más baja y la mayor fidelidad (ver tabla adjunta). Además de la selección de enzimas, los investigadores pueden reducir aún más la tasa de mutación de la PCR optimizando las condiciones de reacción, incluida la optimización de la composición del tampón, la concentración de polimerasa termoestable y la optimización del número de ciclos de PCR.
Categorías de productos
POR QUÉ ELEGIRNOS
Desde su establecimiento, nuestra fábrica ha estado desarrollando productos de primera clase mundial con la adhesión al principio
de calidad primero. Nuestros productos han ganado una excelente reputación en la industria y son valiosos y confiables entre los clientes nuevos y antiguos.








