Mezcla de amplificación de alta fidelidad TIANSeq
Río abajo
Amplificación por PCR de la biblioteca NGS, amplificación por PCR de secuenciación de primera generación, clonación de alta fidelidad, detección de SNP, mutación específica del sitio, etc.
Características
■ Amplificación de alta eficiencia: asegura la tasa de conversión y reduce los ciclos de amplificación.
■ Preferencia baja: eficiencia de amplificación equilibrada para plantillas de ADN con diferentes contenidos de% de GC.
■ Alta especificidad: con propiedad HotStart y fuerte especificidad.
■ Alta fidelidad: la fidelidad es 50 veces mayor que la ADN polimerasa Taq.
■ Alta sensibilidad: la entrada de plantilla puede ser tan baja como 1 pág.
Todos los productos se pueden personalizar para ODM / OEM. Para detalles,haga clic en Servicio personalizado (ODM / OEM)
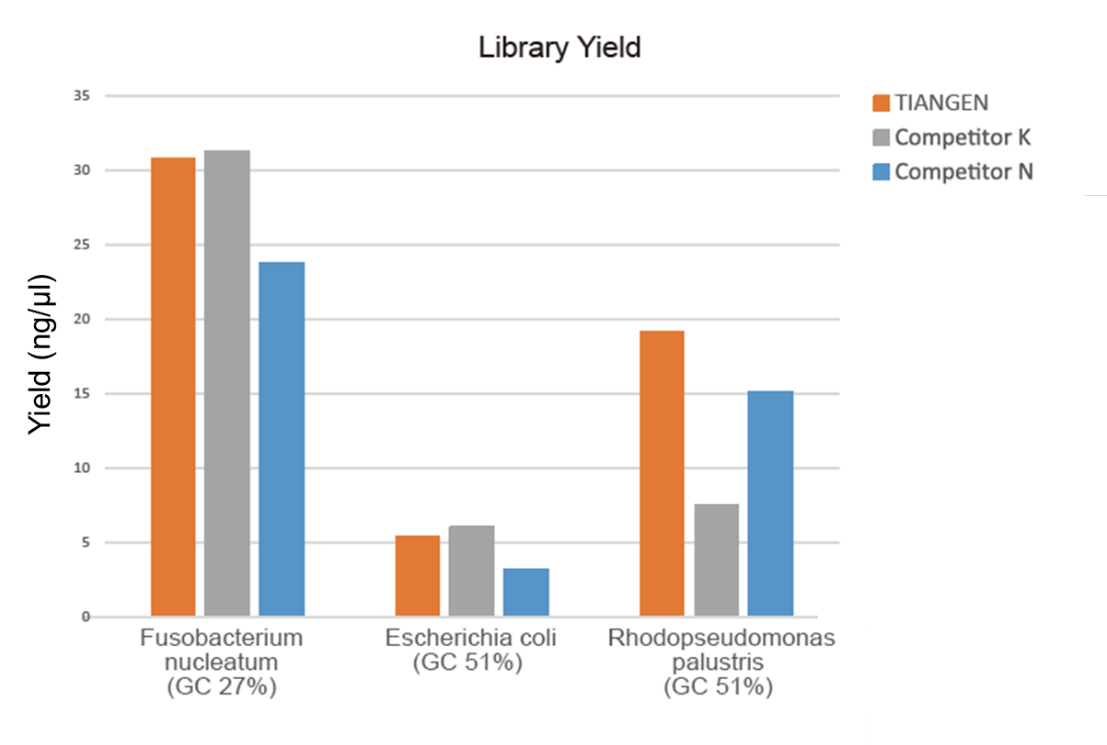 |
Figura 1. El enriquecimiento de la biblioteca de ADN genómico con diferentes proporciones de GC (entrada genómica 10 ng, amplificación de 8 ciclos) se llevó a cabo simultáneamente mediante el uso de TIANSeq HiFi Amplification Mix y la enzima HiFi del proveedor K y N, y Agilent detectó el rendimiento de la biblioteca. 2100. Los resultados mostraron que TIANSeq HiFi Amplification Mix tiene un alto rendimiento de biblioteca, con una fuerte universalidad para diferentes contenidos de GC, y el rendimiento de enriquecimiento de la biblioteca fue mejor que el de otros proveedores. |
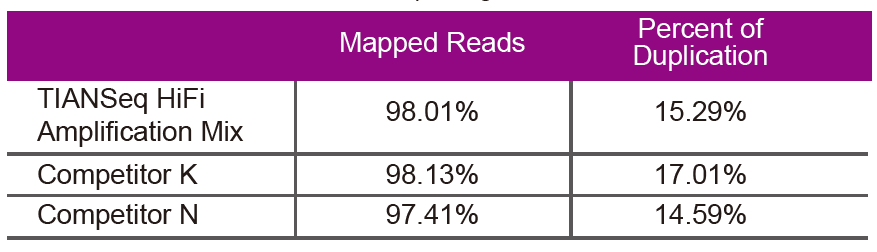 |
Secuencia de datosUtilice la mezcla de amplificación de alta fidelidad TIANSeq y la enzima de alta fidelidad especialmente utilizada para la amplificación de la biblioteca NGS del proveedor K y N para la amplificación de la biblioteca del mismo ADN genómico (la entrada del genoma es de 10 ng). Después de la secuenciación, analice la cobertura y la tasa de duplicación de la biblioteca. |
Preferencia de GC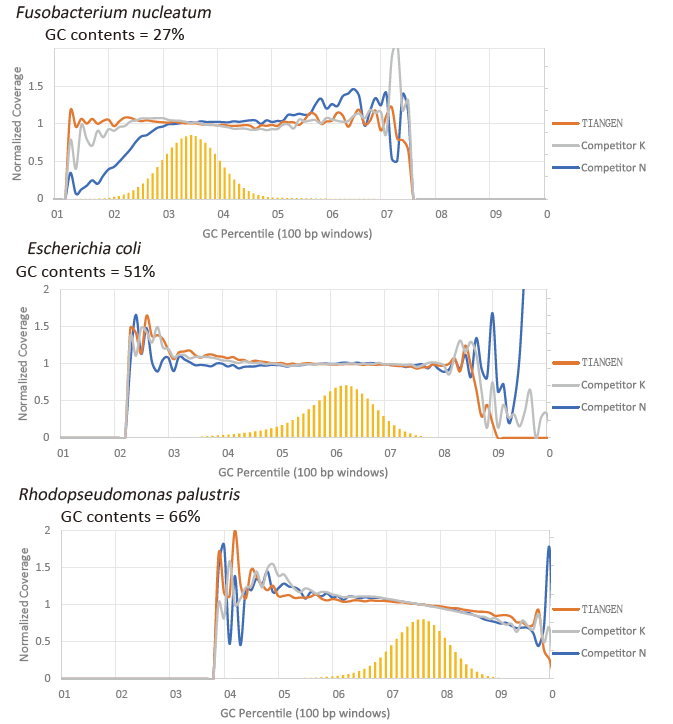 |
Figura 2. Amplifique las bibliotecas de genomas con diferentes contenidos de CG utilizando TIANSeq HiFi Amplification Mix y el HIFi del proveedor K y N. El resultado muestra que la uniformidad de la biblioteca de amplificación TIANSeq HiFi Amplification Mix es buena y sin preferencia de GC, lo que equivale a los resultados de la empresa K y ligeramente mejor que los productos de la empresa N. Los resultados muestran que la cobertura de la biblioteca de amplificación TIANSeq HiFi Amplification Mix es alta, la tasa de duplicación cumplió con los requisitos y el rendimiento de amplificación de la biblioteca es equivalente al de la competencia. |
En la actualidad, la tecnología de secuenciación de alto rendimiento se basa principalmente en la tecnología de secuenciación de próxima generación. Como la longitud de lectura de la tecnología de secuenciación de próxima generación es limitada, debemos dividir la secuencia de longitud completa en pequeñas bibliotecas de fragmentos para secuenciar. De acuerdo con las necesidades de los diferentes experimentos de secuenciación, solemos elegir la secuenciación de un solo extremo o la secuenciación de dos extremos. Actualmente, los fragmentos de ADN de la biblioteca de secuenciación de próxima generación se distribuyen generalmente en el intervalo de 200 a 800 pb.

a) El ADN es de mala calidad y contiene inhibidores. Utilice muestras de ADN de alta calidad para evitar la inhibición de la actividad enzimática.
b) La cantidad de muestra de ADN es insuficiente cuando se utiliza un método sin PCR para construir la biblioteca de ADN. Cuando la entrada del ADN fragmentado supera los 50 ng, el flujo de trabajo libre de PCR se puede llevar a cabo de forma selectiva durante el proceso de construcción de la biblioteca. Si el número de copias de la biblioteca es demasiado bajo para secuenciarlo directamente, la biblioteca de ADN se puede amplificar mediante PCR después de la ligación del adaptador.
c) La contaminación por ARN conduce a una cuantificación inicial inexacta del ADN. Puede existir contaminación por ARN en el proceso de purificación del ADN genómico, lo que puede dar lugar a una cuantificación inexacta del ADN y una carga insuficiente del ADN durante la construcción de la biblioteca. El ARN se puede eliminar mediante el tratamiento con ARNasa.
A-1
a) Aparecen fragmentos pequeños (60 pb-120 pb) Los fragmentos pequeños suelen ser fragmentos adaptadores o dímeros formados por adaptadores. La purificación con perlas magnéticas Agencourt AMPure XP puede eliminar eficazmente estos fragmentos de adaptador y garantizar la calidad de la secuenciación.
b) Aparecen fragmentos grandes en la biblioteca después de la amplificación por PCR. El tamaño del fragmento de ADN de la biblioteca aumentará en 120 pb después de que se ligue el adaptador. Si el fragmento de ADN aumenta en más de 120 pb después de la ligadura del adaptador, podría deberse a una amplificación anormal del fragmento de una amplificación por PCR excesiva. Reducir el número de ciclos de PCR puede prevenir la situación.
c) Tamaño anormal de los fragmentos de ADN de la biblioteca después de la ligadura del adaptador La longitud del adaptador en este kit es de 60 pb. Cuando los dos extremos del fragmento se ligan a los adaptadores, la longitud solo aumentará en 120 pb. Cuando utilice un adaptador que no sea el proporcionado por este kit, comuníquese con el proveedor para proporcionar información relevante, como la longitud del adaptador. Asegúrese de que el flujo de trabajo y la operación del experimento sigan los pasos descritos en el manual.
d) Tamaño anormal del fragmento de ADN antes de la ligadura del adaptador El motivo de este problema puede deberse a condiciones de reacción incorrectas durante la fragmentación del ADN. Deben usarse diferentes tiempos de reacción para diferentes entradas de ADN. Si la entrada de ADN es superior a 10 ng, recomendamos elegir el tiempo de reacción de 12 min como tiempo de inicio para la optimización, y el tamaño del fragmento producido en este momento está principalmente en el rango de 300-500 pb. Los usuarios pueden aumentar o disminuir la longitud de los fragmentos de ADN durante 2-4 minutos según sus propios requisitos para optimizar los fragmentos de ADN con el tamaño requerido.
A-2
a) El tiempo de fragmentación no está optimizado Si el ADN fragmentado es demasiado pequeño o demasiado grande, consulte las Directrices para la selección del tiempo de fragmentación que se proporcionan en las instrucciones para determinar el tiempo de reacción y utilice este punto de tiempo como control, además configure un sistema de reacción para prolongar o acortar 3 min para hacer un ajuste más preciso en el tiempo de fragmentación.
A-3
Distribución anormal del tamaño del ADN después del tratamiento de fragmentación
a) Método de descongelación incorrecto del reactivo de fragmentación, o el reactivo no se mezcla completamente después de la descongelación. Descongele el reactivo 5 × Fragmentation Enzyme Mix en hielo. Una vez descongelado, mezcle el reactivo uniformemente moviendo suavemente la parte inferior del tubo. ¡No agite el reactivo en el vórtex!
b) La muestra de entrada de ADN contiene EDTA u otros contaminantes El agotamiento de iones de sal y agentes quelantes en la etapa de purificación del ADN es particularmente importante para el éxito del experimento. Si el ADN se disuelve en 1 × TE, utilice el método proporcionado en las instrucciones para realizar la fragmentación. Si la concentración de EDTA en la solución es incierta, se recomienda purificar el ADN y disolverlo en agua desionizada para su posterior reacción.
c) Cuantificación inicial inexacta del ADN El tamaño del ADN fragmentado está estrechamente relacionado con la cantidad de ADN introducido. Antes del tratamiento de fragmentación, la cuantificación precisa del ADN utilizando Qubit, Picogreen y otros métodos es esencial para determinar la cantidad exacta de ADN en el sistema de reacción.
d) La preparación del sistema de reacción no sigue las instrucciones. La preparación del sistema de reacción fragmentado debe realizarse en hielo estrictamente de acuerdo con las instrucciones. Para asegurar el mejor efecto, todos los componentes de la reacción deben colocarse en hielo y la preparación del sistema de reacción debe llevarse a cabo después del enfriamiento completo. Una vez completada la preparación, mueva o pipetee para mezclar bien. ¡No vórtice!
1. Un método de mezcla inadecuado (vórtice, oscilación violenta, etc.) provocará una distribución anormal de los fragmentos de la biblioteca (como se muestra en la siguiente figura), lo que afectará la calidad de la biblioteca. Por lo tanto, cuando prepare la solución de reacción de Fragmentation Mix, pipetee suavemente hacia arriba y hacia abajo para mezclar, o use la yema del dedo para mover y mezclar uniformemente. Tenga cuidado de no mezclar con vórtice.

2. Se debe utilizar ADN de alta pureza para la construcción de la biblioteca.
■ Buena integridad del ADN: la banda de electroforesis tiene más de 30 kb, sin colas
■ OD260 / 230:> 1,5
■ OD260 / 280: 1,7-1,9
3. La cantidad de entrada de ADN debe ser precisa. Se sugiere utilizar los métodos Qubit y PicoGreen para cuantificar el ADN, en lugar de Nanodrop.
4. Debe determinarse el contenido de EDTA en la solución de ADN. El EDTA tiene una gran influencia en la reacción de fragmentación. Si el contenido de EDTA es alto, la purificación del ADN debe realizarse antes de la prueba posterior.
5. La solución de reacción de fragmentación debe prepararse en hielo. El proceso de fragmentación es sensible a la temperatura y al tiempo de reacción (especialmente después de añadir potenciador). Para garantizar la precisión del tiempo de reacción, prepare el sistema de reacción en hielo.
6. El tiempo de reacción de fragmentación debe ser preciso. El tiempo de reacción del paso de fragmentación afectará directamente el tamaño de los productos del fragmento, lo que afectará a la distribución de tamaño de los fragmentos de ADN en la biblioteca.
1. ¿Qué tipo de muestra es aplicable a este kit?
El tipo de muestra aplicable de este kit puede ser ARN total o ARNm purificado con buena integridad del ARN. Si se utiliza ARN total para construir la biblioteca, se recomienda utilizar el kit de reducción de ARNr (nº de cat. 4992363/4992364/4992391) para eliminar primero el ARNr.
2. ¿Se pueden utilizar muestras de FFPE para construir una biblioteca con este kit?
El ARNm en las muestras de FFPE se degradará hasta cierto punto, con una integridad relativamente pobre. Al utilizar este kit para la construcción de la biblioteca, se recomienda optimizar el tiempo de fragmentación (acortar el tiempo de fragmentación o no realizar la fragmentación).
3. Usando el paso de selección de tamaño provisto en el manual del producto, ¿qué puede causar que el segmento insertado parezca una ligera desviación?
La selección del tamaño se llevará a cabo estrictamente de acuerdo con el paso de selección del tamaño en este manual del producto. Si hay una desviación, la razón podría ser que las perlas magnéticas no están equilibradas a temperatura ambiente o no están completamente mezcladas, la pipeta no es precisa o el líquido permanece en la punta. Se recomienda utilizar las puntas con baja adsorción para el experimento.
4. Selección de adaptadores en la construcción de bibliotecas
El kit de construcción de la biblioteca no contiene reactivo adaptador y se recomienda utilizar este kit junto con el adaptador de índice único TIANSeq (Illumina) (4992641/4992642/4992378).
5. Control de calidad de la biblioteca
Detección cuantitativa de la biblioteca: Qubit y qPCR se utilizan para determinar la concentración de masa y la concentración molar de la biblioteca, respectivamente. La operación es estrictamente de acuerdo con el manual del producto. La concentración de la biblioteca generalmente cumplirá los requisitos de secuenciación NGS. Detección del rango de distribución de la biblioteca: uso del bioanalizador Agilent 2100 para detectar el rango de distribución de la biblioteca.
6. Selección del número de ciclo de amplificación
De acuerdo con las instrucciones, el número de ciclos de PCR es de 6 a 12, y el número de ciclos de PCR necesarios debe seleccionarse de acuerdo con la entrada de la muestra. En bibliotecas de alto rendimiento, la sobreamplificación suele producirse en diversos grados, lo que se manifiesta por un pico ligeramente mayor después del pico del rango objetivo en la detección del bioanalizador Agilent 2100, o la concentración detectada de Qubit es menor que la de qPCR. La sobre amplificación leve es un fenómeno normal, que no afecta la secuenciación de la biblioteca y el análisis de datos posterior.
7. Aparecen picos en el perfil de detección del bioanalizador Agilent 2100
La aparición de picos en la detección del bioanalizador Agilent 2100 se debe a la fragmentación desigual de las muestras, donde habrá más fragmentos de cierto tamaño, y esto se hará más obvio después del enriquecimiento por PCR. En este caso, se sugiere no realizar la selección de tamaño, es decir, establecer la condición de fragmentación a 94 ° C durante 15 min de incubación, donde la distribución de los fragmentos es pequeña y concentrada, y se puede mejorar la homogeneidad.
Categorías de productos
POR QUÉ ELEGIRNOS
Desde su establecimiento, nuestra fábrica ha estado desarrollando productos de primera clase mundial con la adhesión al principio
de calidad primero. Nuestros productos han ganado una excelente reputación en la industria y son valiosos y confiables entre los clientes nuevos y antiguos.








