FastKing gDNA Disipador RT SuperMix
Características
■ Rápido: un paso para completar la eliminación del genoma y la transcripción inversa eficiente en 18 minutos con solo agregar plantillas.
■ Alta eficiencia: la transcriptasa inversa se modifica con un motivo hidrofóbico, con una eficiencia de RT superior al 95%.
■ Simple y fácil: la exclusiva DNasa termosensible tiene un efecto rápido, alta eficiencia con un tiempo de reacción más corto y no afectará al ADNc.
Especificación
Escribe: Transcriptasa inversa genéticamente modificada, gDNase
Procedimientos: Un paso (eliminación de ADN genómico y RT)
Eficiencia RT: > 95%
Plantilla: 1 ng- 2 μg
Tiempo de operacion: ~ 18 min
Aplicaciones: El ADNc de transcripción inversa se puede utilizar en PCR convencional, PCR en tiempo real, construcción de bibliotecas de ADNc, SAGE (Análisis en serie de expresión génica), extensión de cebadores y otros experimentos convencionales.
Todos los productos se pueden personalizar para ODM / OEM. Para detalles,haga clic en Servicio personalizado (ODM / OEM)
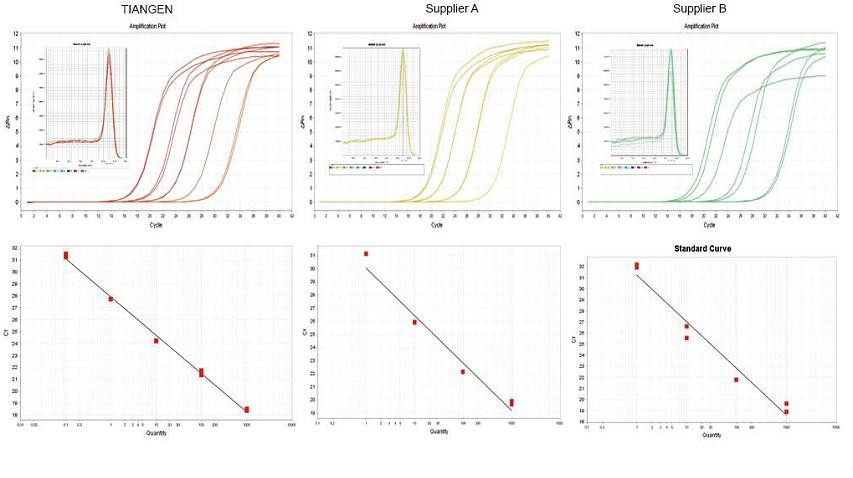 |
Ejemplo experimental 1. Se sintetizó ADNc usando reactivos cuantitativos inversos de un paso de TIANGEN FastKing gDNA Dispelling RT SuperMix, productos relevantes del Proveedor A y del Proveedor B respectivamente. Detectar el gen RN5 de ratones usando TIANGEN Talent qPCR PreMix (SYBR Green), y se analizaron la curva de amplificación, la curva de fusión y la curva estándar. Los resultados muestran que TIANGEN FastKing gDNA Dispelling RT SuperMix tiene el valor Ct cuantitativo más alto después de la transcripción inversa y una excelente resistencia al estrés, y tiene ventajas obvias para las plantillas con altos residuos de impurezas. |
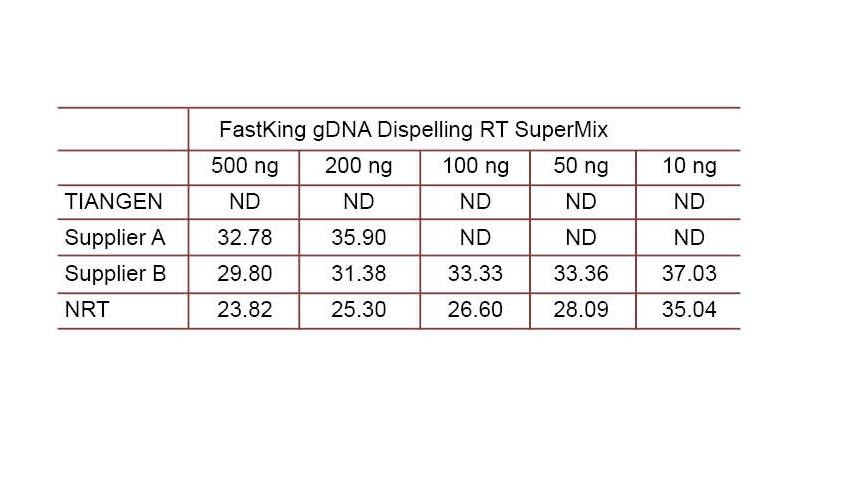 |
Ejemplo experimental 2. Se sintetizó ADNc usando reactivos cuantitativos inversos de un paso de TIANGEN FastKing gDNA Dispelling RT SuperMix, productos relevantes del Proveedor A y del Proveedor B, respectivamente. Detecte el gen HsG humano usando TIANGEN Talent qPCR PreMix (SYBR Green) y agregue manualmente diferentes concentraciones de ADN genómico para detectar la capacidad de eliminación de ADNg de diferentes reactivos. Los resultados de CT muestran que TIANGEN FastKing gDNA Dispelling RT SuperMix tiene una excelente capacidad para eliminar el ADN genómico. Se pueden eliminar perfectamente hasta 500 ng de residuos de ADN genómico sin afectar los resultados. ND: No detectado. NRT: Detección de la mezcla sin transcripción inversa. |
El ARN A-1 se degrada
——Purificar ARN de alta calidad sin contaminación. El material del que se extrae el ARN debe ser lo más fresco posible para evitar la degradación del ARN. Analice la integridad del ARN en gel desnaturalizado antes de la reacción de RT. Después de la extracción de ARN, debe almacenarse en formamida al 100%. Si se usa un inhibidor de ARNasa, la temperatura de calentamiento debe ser <45 ° C y el pH debe ser menor de 8.0; de lo contrario, el inhibidor liberará toda la ARNasa unida. Además, el inhibidor de RNasa debe agregarse en soluciones que contengan DTT ≥ 0,8 mM.
El ARN A-2 contiene inhibidores de las reacciones de transcripción inversa.
——Los inhibidores de la transcripción inversa incluyen SDS, EDTA, glicerol, pirofosfato de sodio, espermidina, formamida, sal de guanidina, etc. Mezcle el ARN de control con la muestra y compare el rendimiento con la reacción del ARN de control para comprobar si hay un inhibidor. Lavar la precipitación de ARN con etanol al 70% (v / v) para eliminar los inhibidores.
A-3 Hibridación insuficiente de cebadores utilizados para sintetizar la primera hebra de ADNc
——Determine que la temperatura de hibridación es adecuada para los cebadores utilizados en el experimento. Para hexámeros aleatorios, se recomienda mantener la temperatura a 25 ° C durante 10 minutos antes de alcanzar la temperatura de reacción. Para cebadores de genes específicos (GSP), pruebe con otro GSP o cambie a oligo (dT) o hexámero aleatorio.
A-4 Pequeña cantidad de ARN inicial
——Aumentar la cantidad de ARN. Para muestras de ARN de menos de 50 ng, se pueden usar 0,1 μg a 0,5 μg de acetil BSA en la síntesis de la primera hebra de ADNc
A-5 La secuencia diana no se expresa en los tejidos analizados.
——Pruebe con otros pañuelos.
La reacción de PCR A-6 falla
——Para RT-PCR de dos pasos, la plantilla de ADNc en el paso de PCR no puede exceder 1/5 del volumen de reacción.
A-1 Recolección no específica de cebadores y plantillas
——El extremo 3 'de los cebadores no debe contener 2-3 dG o dC. Utilice cebadores específicos de genes en la síntesis de la primera hebra en lugar de cebadores aleatorios u oligo (dT). Utilice una temperatura de recocido más alta en los primeros ciclos y luego una temperatura de recocido más baja. Utilice la polimerasa de ADN Taq de inicio en caliente para PCR para mejorar la especificidad de la reacción.
A-2 Diseño deficiente de cebadores específicos de genes
——Siga los mismos principios para el diseño de cebadores de amplificación.
ARN A-3 contaminado con ADN genómico
——Tratar el ARN con ADNasa I de grado PCR. Configure una reacción de control sin transcripción inversa para detectar la contaminación del ADN.
A-4 Formación de dímero de imprimación
——Diseñar cebadores sin secuencias complementarias en el extremo 3 '.
A-5 Mg demasiado alto2+ concentración
——Optimizar Mg2+ concentración para cada combinación de plantilla y cebador
A-6 Contaminado con ADN extraño
——Utilice puntas resistentes a aerosoles y enzimas UDG.
A-1 El contenido del producto de la primera hebra es demasiado alto
——Reducir la cantidad del producto de la primera hebra en el paso de reacción de PCR convencional.
A-2 Cantidad de cebador demasiado alta en la reacción de PCR
——Reducir la entrada del cebador.
A-3 Demasiados ciclos
——Optimizar las condiciones de la reacción de PCR y reducir el número de ciclos de PCR.
A-4 Temperatura de recocido demasiado baja
——Aumentar la temperatura de recocido para evitar la iniciación y extensión inespecíficas.
A-5 Amplificación no específica de fragmentos de oligonucleótidos generados por la degradación del ADN por ADNasa ——Extraiga ARN de alta calidad para evitar la contaminación del ADN.
La RT-PCR consiste en realizar la transcripción inversa del ARN en ADNc y luego utilizar el ADNc transcrito en reversa como plantilla para la reacción de PCR para amplificar el fragmento diana. Elija cebadores aleatorios, Oligo dT y cebadores específicos de genes de acuerdo con las condiciones específicas del experimento. Todos los cebadores anteriores pueden usarse para ARNm de células eucariotas cortas sin estructura de horquilla.
Primer aleatorio: Adecuado para ARN largo con estructura de horquilla, así como todo tipo de ARN como ARNr, ARNm, ARNt, etc. Se utilizan principalmente para la reacción RT-PCR de plantilla única.
Oligo dT: Adecuado para ARN con colas de PolyA (ARN procariótico, oligo dT rRNA eucariótico y tRNA no tienen colas de PolyA). Debido a que Oligo dT está unido a la cola de PolyA, se requiere que la calidad de las muestras de ARN sea alta, e incluso una pequeña cantidad de degradación reducirá en gran medida la cantidad de síntesis de ADNc de longitud completa.
Cebador específico de gen: complementario a la secuencia molde, adecuado para situaciones en las que se conoce la secuencia diana.
Hay dos maneras:
1. Método de referencia interno: en teoría, el ADNc son fragmentos de ADN de diferentes longitudes, por lo que el resultado de la electroforesis es un frotis. Si la abundancia de ARN es baja, no se mostrará ningún producto en la electroforesis, pero esto no significa que ningún producto será amplificado por PCR. En general, se puede usar una referencia interna para detectar ADNc. Si la referencia interna tiene resultados, la calidad del cDNA se puede garantizar básicamente (en algunos casos, si el fragmento del gen diana es demasiado largo, puede haber excepciones).
2. Si hay un gen conocido amplificado por esta plantilla, puede ser verificado por los cebadores de este gen. La amplificación de la referencia interna no significa necesariamente que no haya ningún problema con el ADNc. Debido a que la referencia interna tiene una gran abundancia de ADNc, es fácil de amplificar. Si el ADNc se degrada parcialmente por diversas razones, desde la perspectiva de la probabilidad, los resultados de la PCR de genes diana de baja abundancia se verán muy afectados. Si bien la referencia interna todavía es alta en abundancia, la amplificación probablemente no se verá afectada.
Degrada parcialmente el ARN. Detecta la integridad y purifica el ARN.
El contenido de ARN de diferentes especies puede ser diferente, pero en general, el ARN total extraído debe contener dos bandas claras 28S y 18S en electroforesis en gel, y el brillo de la primera banda debe ser dos veces mayor que el de la última. La banda 5S indica que el ARN se ha degradado y su brillo es proporcional al grado de degradación. La amplificación exitosa de la referencia interna no significa que no haya ningún problema con el ARN, porque la referencia interna es muy abundante, el ARN puede amplificarse siempre que la degradación no sea grave. El OD260/SOBREDOSIS280La proporción de ARN puro medido por espectrofotómetro debe estar entre 1,9 y 2,1. Una pequeña cantidad de impureza proteica en el ARN reducirá la proporción. Siempre que el valor no sea demasiado bajo, RT no se verá afectado. Lo que más importa para RT es la integridad del ARN.
La extensión del gen de referencia interno solo puede indicar que la RT ha tenido éxito, pero no está necesariamente relacionada con la calidad de la cadena de ADNc. Debido a que los fragmentos de referencia internos son generalmente de tamaño pequeño y de alta expresión, es más fácil tener éxito en la transcripción inversa. Sin embargo, el tamaño y la expresión del gen diana varía de un gen a otro. La calidad del cDNA no se puede juzgar solo por referencia interna, especialmente para los fragmentos diana de más de 2 kb.
Algunas muestras tienen estructuras secundarias complejas, tienen un rico contenido de GC o son valiosas con poca abundancia. En estos casos, se debe seleccionar la transcriptasa inversa apropiada de acuerdo con el tamaño del fragmento diana y la muestra. Para las plantillas de ARN con alto contenido de GC y estructura secundaria compleja, es difícil abrir la estructura secundaria a baja temperatura o con transcriptasa inversa común. Para estas plantillas, se puede seleccionar Quant Reverse Transcriptase, ya que su rendimiento de transcripción inversa es obviamente mejor que el de la transcriptasa inversa de la serie M-MLV, que puede realizar la transcripción inversa de varias plantillas de ARN de manera eficiente y transcribir ARN en la primera hebra de ADNc en la máxima extensión. Cuando se utiliza un kit de transcriptasa inversa general, el sistema de 20 μl solo puede transcribir de manera efectiva 1 μg de ARN total. Preste atención a la capacidad máxima de RT del kit. Si la plantilla se agrega en exceso, la transcripción inversa favorecerá al ARN con alta abundancia. Por lo tanto, es mejor no exceder la capacidad máxima del sistema.
A-1 Determinar si el ARN se degrada severamente y si la RT tiene éxito
En general, el motivo del fallo de la amplificación de referencia interna a menudo se debe a una degradación grave del ARN. Otra posible razón es la falla de la transcripción inversa. La referencia interna no se puede utilizar como estándar para juzgar la calidad del ADNc monocatenario, pero se puede utilizar como estándar para juzgar si la transcripción inversa es exitosa si no hay ningún problema con la calidad del ARN. Lo más importante en el proceso de transcripción inversa es mantener una temperatura constante y un sistema de reacción constante para mejorar la eficiencia de la reacción.
A-2 Determine si los cebadores para amplificar los genes de referencia internos son fiables y si hay algún problema con los reactivos utilizados en la PCR.
Para la cuantificación relativa, el ARN debe cuantificarse antes de la transcripción inversa, que también se requiere en muchos kits de transcripción inversa, por ejemplo, cuantificar la entrada de ARN como 1 μg. Dado que el ADNc de transcripción inversa es una solución mixta, que incluye ARN, oligo dT, enzima, dNTP e incluso un pequeño residuo de ADN, se producirá una desviación, por lo que es imposible cuantificar con precisión el ADNc. Por tanto, es necesaria la cuantificación del ARN. Teniendo en cuenta que la eficiencia de la transcripción inversa es la misma entre diferentes muestras, la cantidad de ADNc obtenido debe ser la misma y el análisis cuantitativo puede mostrar la comparación de los niveles de expresión de diferentes genes en la misma cantidad de ARN total. Cuando se realiza una PCR cuantitativa de fluorescencia relativa, es posible que no se requiera ADNc cuantitativo después de la transcripción inversa porque el gen de referencia interno puede actuar como referencia.
Está principalmente relacionado con los genes y la transcripción inversa de fragmentos largos no es factible para la mayoría de los genes. En primer lugar, la eficacia de la transcripción inversa es mucho menor que la de la PCR. En segundo lugar, la región rica en GC y la estructura secundaria de muchos genes restringen tanto la transcripción inversa como la PCR. Finalmente, la fidelidad y la eficiencia de amplificación de la PCR son difíciles de garantizar al mismo tiempo. En el proceso de transcripción inversa, nadie puede garantizar la obtención de un fragmento largo para genes de copia baja, especialmente si se utiliza oligo dT. En cuanto a 5 'UTR con más GC, es aún más difícil. Por lo tanto, sigue siendo un método razonable invertir la transcripción con cebadores aleatorios, encontrar los sitios de escisión naturales en el fragmento diana, amplificar por segmentos y luego realizar la digestión de restricción y la ligación. En general, es difícil amplificar directamente fragmentos mayores de 2 kb, pero no siempre es imposible obtener: 1.En primer lugar, garantizar la integridad del ARN / ARNm y se prefiere la extracción con TRIZOL. 2.El kit M-MLV RT-PCR se puede utilizar directamente. Extienda el tiempo de recocido y aumente el número de ciclos en el proceso de amplificación correctamente. Alternativamente, se puede aplicar PCR anidada, o llevar a cabo una o dos reacciones primero con desnaturalización y tiempo de extensión adecuadamente extendidos antes de la amplificación por PCR normal, lo que puede ayudar a extender los fragmentos. Preste atención a la fidelidad de la polimerasa. 3.Long Taq se puede utilizar en PCR para obtener resultados ideales. 4.Para la aplicación de expresión de proteínas, se debe aplicar polimerasa de alta fidelidad.
TIANGEN ofrece dos tipos de transcriptasa inversa: Quant / King RTase y TIANScript M-MLV. La principal diferencia entre ellos es la cantidad de plantillas de entrada. Quant es una transcriptasa inversa única, que es diferente de la M-MLV comúnmente utilizada derivada del virus de la leucemia murina de Moloney. Quant es una nueva transcriptasa inversa de alta eficiencia expresada de forma recombinante mediante ingeniería de Escherichia coli. Quant es adecuado para amplificar 50 ng-2 μg de ARN con alta actividad de transcripción inversa y alto rendimiento. En comparación con el MMLV o AMV ordinario, la característica más importante de Quant es que tiene una afinidad muy fuerte con las plantillas de ARN y puede revertir las plantillas del complejo de transcripción sin desnaturalización a alta temperatura. Para plantillas con mayor contenido de GC, la eficiencia inversa es mayor. Sin embargo, esta transcriptasa inversa tiene actividad RNasa H, que puede afectar la longitud del producto de ADNc (adecuado para plantillas de <4,5 kb). Para la transcripción inversa convencional, se recomienda la transcriptasa inversa TIANScript MMLV. Esta RTasa es una enzima modificada con una actividad de ARNasa H muy débil, que es adecuada para una síntesis de ADNc prolongada (> 5 kb).
La transcripción inversa en un solo paso y la amplificación por PCR se completan en el mismo tubo sin abrir la tapa del tubo entre la síntesis y la amplificación del cDNA, lo que es útil para reducir la contaminación. Dado que todas las muestras de ADNc obtenidas se utilizan para la amplificación, la sensibilidad es mayor, con un mínimo de 0,01 pg de ARN total. Para el éxito de la RTPCR de un paso, los cebadores específicos de genes se utilizan generalmente para iniciar la síntesis de ADNc. El método de dos pasos, a saber, la transcripción inversa y la amplificación por PCR, se lleva a cabo en dos pasos. En primer lugar, se lleva a cabo la transcripción inversa a partir de una plantilla de ARN para obtener ADNc, y el ADNc obtenido se somete a una o más reacciones de PCR diferentes. El método de dos pasos puede utilizar oligo (dT) o cebadores aleatorios para guiar la síntesis de la primera hebra de cDNA, y puede realizar una transcripción inversa de toda la información de mRNA de una muestra específica.
Categorías de productos
POR QUÉ ELEGIRNOS
Desde su establecimiento, nuestra fábrica ha estado desarrollando productos de primera clase mundial con la adhesión al principio
de calidad primero. Nuestros productos han ganado una excelente reputación en la industria y son valiosos y confiables entre los clientes nuevos y antiguos.